PyAuDiSam
Il s'agit à la fois d'un programme en ligne de commande et d'un module Python 3 permettant d'automatiser massivement de multiples variantes d'analyse en Distance Sampling d'inventaires naturalistes de terrain avec relevé des distances, et de dépouiller facilement la masse des résultats qui en découle, grâce à l'automatisation des étapes, règles et heuristiques habituellement mises en oeuvre par un analyste humain.
La version 1.0.1 a été publiée en mai 2023 :
- sur github.com : https://github.com/denmedius/pyaudisam,
- sur pypi.org : https://pypi.org/project/pyaudisam.
Par rapport au logiciel interactif "Distance", l'intérêt principal est de simplifier considérablement le paramétrage des analyses et la gestion des données d'inventaire, et ainsi de limiter beaucoup les erreurs possibles de saisie et incohérences "d'étourderie" qui arrivent inévitablement quand on est noyé par la quantité de travail (avec Distance, 10 analyses, c'est facile, mais 1000 ... c'est une autre histoire).
Un peu plus en détails :
- une fois les données de l'inventaire mises au propre (individualisées ou en "clusters", en mode line ou point transect), via le module "visionat" par exemple (à publier aussi),
- une pré-analyse par échantillon est lancée de manière à prendre le poul de la qualité des données et de ce qui peut en être espéré a priori => un rapport HTML et ou Excel est produit, la version HTML permettant en plus de consulter tous les résultats détaillés produits par le moteur MCDS sous-jacent ;
- il est temps ensuite de définir (à la main) les analyses qui devront être lancées pour chaque échantillon : on le fait via un classeur Excel ou Libre Office multi-onglet où les combinaisons d'échantillons et de paramètres d'analyse (modèles, critères, ...) sont spécifiées de manière explicite (une par une) ou implicite (combinatoire),
- parmi ces paramètres, les troncatures en distance à gauche et à droite peuvent être spécifiées "automatiques" ce qui activera leur détermination automatique (via un algorithme d'optimisation assez lourd) ; cela allonge beaucoup les calculs (qq secondes => qq heures), mais permet d'explorer sans fatigue des milliers de valeurs de paramètres, nombre inaccessible à la main avec le logiciel Distance par exemple,
- on peut alors lancer les analyses ainsi spécifiées, et laisser faire la machine,
- quand les calculs sont terminés, des rapports d'analyse HTML et / ou Excel peuvent-être facilement produits de manière à pouvoir consulter simplement l'ensemble des détails de tous les résultats d'analyse, ou à loisir simplement un extrait synthétique,
- le type de rapport le plus utile est celui qui sélectionne automatiquement (critères paramétrables) les "meilleures" analyses à conserver pour chaque échantillon (la / les "meilleures") : l'algorithme sélectionne les 5, 10, 12, 15, 20 meilleures selon divers critères combinés ou alternatifs, selon une méthode proche de ce qu'on fait "à la main" avec Distance ; l'utilisateur a cependant le dernier mot pour le choix final de la ou des analyses à retenir pour chaque échantillon, l'automatisme étant très performant (les premiers tests "grandeur nature" laissent espérer au moins 95 % de bon classements), mais pas parfait,
- pour enchaîner toutes ces étapes, une interface utilisateur simple serait nécessaire, mais n'a pas encore été développée (notion de projet / étude, paramétrage simplifié via des modèles / "templates" tous près, importation des fichiers de données "mises au propre" et des spécifications d'analyse, enchaînement des étapes 1 à 1 ... jusqu'à la production des rapports finaux) ; en attendant cette amélioration d'ergonomie, la ligne de commande de pyaudisam permet tout de même d'effectuer cet enchainement (Cf. documentation) ; et un notebook python est également fourni en exemple pour montrer comment effectuer cet enchaînement via l'API python de pyaudisam.
Pour encore plus de détails (notamment sur la mise en oeuvre concrète de pyaudisam), se référer à la documentation du projet (essentiellement en Anglais), sur github.com ou pypi.org.
Cartographie
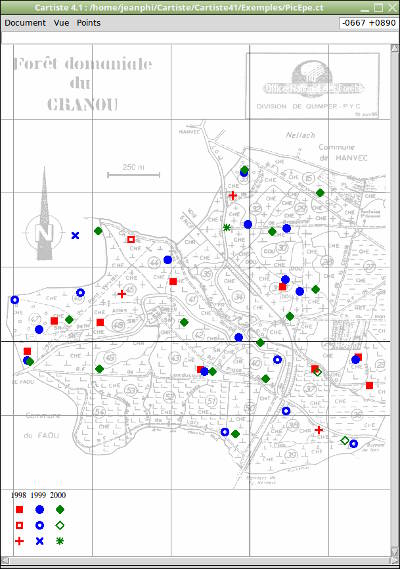
Cartiste est un petit logiciel de cartographie sans prétention que j'ai écrit dans les années 1990 avec le langage Python et le vieux toolkit graphique Tk.
Il permet de (dé)placer comme on veut des symboles graphiques paramétrables (en forme, couleur, orientation, étiquette textuelle, ... etc) sur un fond de carte numérisé, d'afficher leur position selon une échelle réglable, de zoomer, de faire une impression PostScript (et PDF) du résultat, d'importer et d'exporter les points et données libres associées au format CSV.
Son format de document textuel permet de pallier certains petits manques d'ergonomie de l'interface : on peut ainsi modifier / générer les points à la main avec un simple éditeur de texte, et même automatiser ces modifications ou cette génération (en plus du système d'import / export CSV).
Pour l'installer, le lancer et l'utiliser :
- télécharger cette archive 7-zip,
- extraire son contenu sur votre disque dur (avec 7-Zip, par exemple),
- installer Python 2.x si ce n'est pas déjà fait (N.B. Et pas une version 3.x, le portage reste à faire ...)
- adapter puis lancer cartiste.sh ou Cartiste.bat, selon que vous êtes sous Linux ou Windows,
- lire le manuel Cartiste.odt (avec Libre Office 4.x ou 5.x, MS Word lui ne sait pas faire).
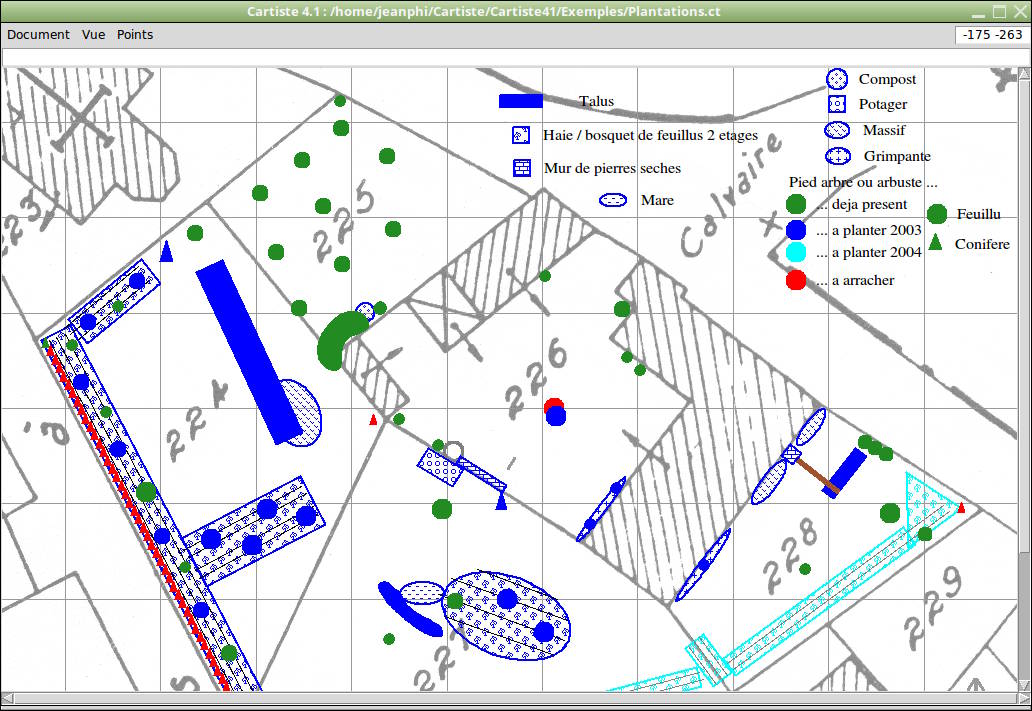
Exemple de plan d'aménagement de mon jardin sur fond cadastral (cliquer pour voir les données) :

Redressement de données ornithologiques de terrain
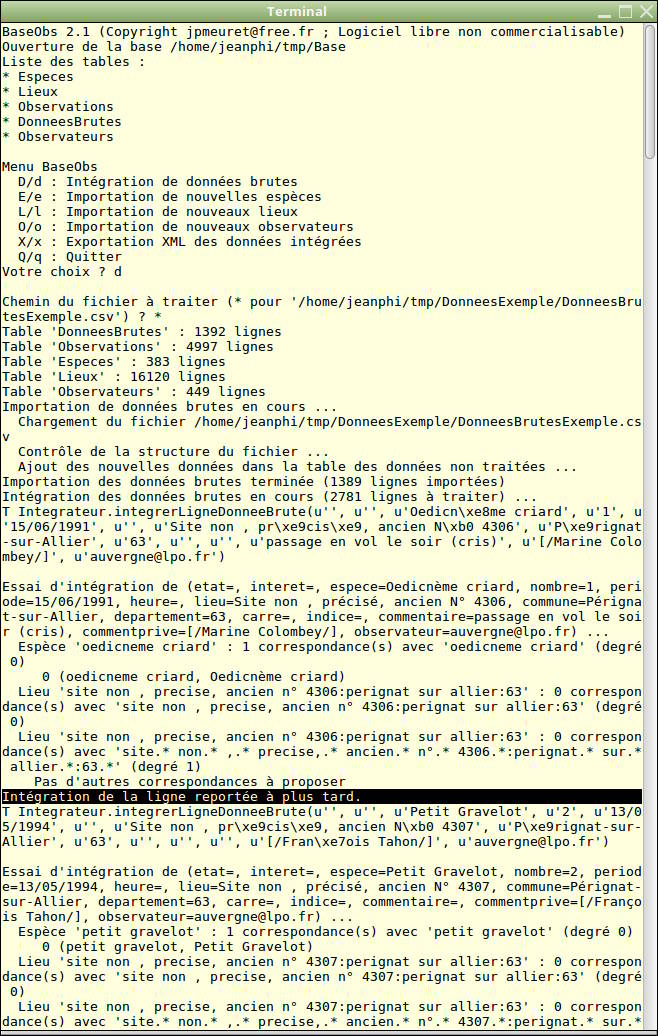
BaseObs est un logiciel écrit en Python 2.x permettant d'intégrer des données ornithologiques "brutes" dans une base de données relationnelle quelconque (en passant par le format CSV), et d'exporter le résultat au format XML d'import massif des sites Visio Nature et Natura List (construits par Biolovision SARL pour la Ligue pour la Protection des Oiseaux et le Muséum National d'Histoire Naturelle).
Il n'a pas d'interface graphique : il fonctionne en mode commande, dans une console, via un menu textuel "à l'ancienne" :-)
Sa particularité essentielle est le support d'abbreviations et d'approximations dans les données brutes pour désigner les espèces, lieux et observateurs (via un système "d'apprentissage" très simple).
Exemple :
- Espèce : "bus s m" ou "bu sa" correspond à "Busard de la Saint-Martin",
- Lieu-dit : "le mar ora, la mout" correspond à "Le Marais d'Orange, La Moutade".
Cela m'a permis, entre 2001 et 2012, de saisir mes données directement sur le terrain sur un PDA Palm (M505 puis Tungsten E2), en entrant aussi peut de caractères que possible, et de les intégrer régulièrement dans la base de la LPO Auvergne (base MS Access, puis "en ligne" Faune Auvergne Rhone-Alpes) ; et aussi d'intégrer facilement et rapidement dans cette base des centaines de milliers de données d'autres observateurs, données pleines de coquilles, ou référençant des lieux dont l'orthographe est variable selon les cartes.
Avec l'avènement de l'appli. NaturaList pour smart-phone, ce logiciel a perdu de son intérêt, mais vous pourrez peut-être utiliser des parties de son code Python ou des idées simples qui y sont implémentées, en particulier :
- un noyau générique rudimentaire de gestion d'une base de données relationnelle (tables au format CSV, tout en mémoire, colonnes non typées, système de requêtage rudimentaire par expressions régulières, pas d'indexation),
- le système d'intégration de données brutes avec support des approximations sur les valeurs de clef (espèce, observateur, lieux).
Vous trouverez plus de détails techniques dans le document BaseObs.odt fourni.
Pour l'installer :
- télécharger cette archive 7-zip,
- extraire son contenu sur votre disque dur (avec 7-Zip, par exemple),
- installer Python 2.x si ce n'est pas déjà fait (N.B. Et pas une version 3.x, le portage n'est pas terminé ...)
- lire le document BaseObs.odt (avec Libre Office 4.x ou 5.x, MS Word lui ne sait pas faire).
- lire le document 0_LisezMoi.txt.

Vivent les logiciels libres !
Dans mes activités quotidiennes, j'utilise autant que possible des logiciels dits "libres", c'est à dire, selon Wikipedia, des logiciels "dont l'utilisation, l'étude, la modification et la duplication en vue de sa diffusion sont permises, techniquement et légalement".
C'est en effet pour moi la seule manière durable d'accéder librement et gratuitement au code source, pour, si besoin :
- le contrôler, le critiquer et le corriger, sans contraintes commerciales ou politiques (objectifs de qualité, de sécurité, d'honnêteté),
- le partager gratuitement (enseignement, formation, ...),
- s'en servir comme base pour de nouvelles constructions (progrès technique), "libres" à leur tour.
N.B. : Cette gratuité et cette liberté ont en théorie une contre-partie : un devoir moral de contribution à l'oeuvre communautaire. Sinon, c'est au mieux du parasitisme, au pire du pillage.
Et s'il faut un dernier argument : les sciences avancent par la publication des résultats et des raisonnements qui y conduisent ; le logiciel avance par la transparence des codes sources.
Ce site internet a par exemple été construit avec (liste non exhaustive) :
- le système d'exploitation Linux Mint Debian Edition 2,
- le gestionnaire de fenêtres Gnome 3,
- le gestionnaire de bureau Cinnamon,
- l'interpréteur de commandes Bash,
- le navigateur internet Mozilla Firefox,
- l'éditeur d'image GIMP,
- le serveur internet Apache,
- l'éditeur de code source GNU Emacs,
- ... etc.
Bien entendu, en dehors du domaine public, des GPL et des Creative Commons, il existe une pléthore de licences logicielles dites "libres", dont certaines sont plus ou moins douteuses : nous n'entrerons pas ici dans les détails, mais sachez-le. Et de toute manière, l'immense majorité d'entre elles est tout à fait respectable, et présente en tout cas des garanties de "liberté" bien supérieures aux logiciels propriétaires ou à sources fermés, gratuits ou pas.
Utopie ou pas ? A vous de voir.